| Este artigo é patrocinado por: «A sua instituição aqui» |
As enzimas de restrição, também denominadas de endonucleases de restrição, desempenham a função de clivagem (corte) da molécula de DNA em reconhecimento a determinadas sequências de nucleótidos.
Estas enzimas reconhecem sequências de 4 a 8 nucleótidos, os denominados locais de restrição.
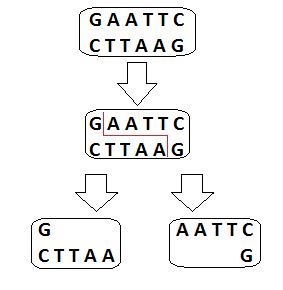
Na figura que se segue é possível verificar a sequência de reconhecimento da enzima de restrição EcoRI, bem como os fragmentos em sequência do corte.
Foram inicialmente descobertas em células bacterianas, relacionadas ao mecanismo de defesa contra DNA exógeno, inativando o organismo invasor a partir da fragmentação do material genético do mesmo.
O nome da enzima tem origem no nome do organismo do qual foi isolada, desta forma:
- EcoRI
E: Escherichia (género)
co: coli (espécie)
R: RY13 (estirpe)
I: ordem de identificação da bactéria
Algumas destas “tesouras moleculares” são ferramentas chave em técnicas de biotecnologia molecular uma vez que cada enzima de restrição, reconhece e fragmenta uma região específica da molécula de DNA, produzindo sempre o mesmo conjunto de fragmentos de DNA. Uma enzima de restrição permite cortar moléculas de DNA de forma controlada e reprodutível.
Desta forma, os fragmentos gerados podem ser submetidos à técnica de eletroforese em gel, e através de análise comparativa dos perfis gerados é possível, por exemplo, a identificação de pessoas (suspeitos de um crime ou determinação da paternidade).
Estas enzimas cortam o DNA em ziguezague deixando extremidades de cadeia única (extremidades coesivas), permitindo o emparelhamento de “caudas” de diferentes fragmentos de DNA cortados com a mesma enzima. Estes fragmentos emparelhados podem depois ser unidos pela enzima DNA ligase com consumo de ATP.
Atualmente, inúmeras enzimas de restrição já foram purificadas de várias espécies de bactérias, a maioria das quais reconhece diferentes sítios de restrição, sendo amplamente comercializadas.
Tipos de enzimas de restrição
A classificação das diferentes enzimas de restrição é baseada na composição da sequência nucleotídica, posição de clivagem, sequência de reconhecimento, cofatores envolvidos e estrutura da enzima em causa:
Tipo I (Impede o seu uso em engenharia genética)
- 3 subunidades: 1 subunidade de restrição (“R”), 1 subunidade de metilação (“M”) e 1 subunidade de reconhecimento (“S”);
- Sequência de reconhecimento é assimétrica;
- Local de hidrólise não é específico e ocorre a mais de 1000 bp de distância;
- Hidrólise é sempre num local diferente e necessita de ATP
Tipo II (Importantes ferramentas em engenharia genética devido à especificidade de corte)
- Duas enzimas: 1 enzima de restrição (Endonuclease) e 1 enzima de metilação (Metilase);
- Estrutura com 4 folhas β e 1 hélice α;
- Sequência de reconhecimento é palindrómica 4 a 8 nucleótidos;
- Hidrólise ocorre dentro ou nas extremidades da sequência reconhecida;
- Não necessita de ATP, apenas Mg2+
Tipo III (Impede o seu uso em engenharia genética)
- 2 subunidades: 1 subunidade de restrição (“R”) e 1 subunidade de metilação e reconhecimento (“MS”);
- Sequência de reconhecimento é assimétrica (5-7 bp);
- Dupla cadeia de DNA hemimetilada;
- Local de hidrólise a mais de 24-26 bp de distância;
- Hidrólise é sempre num local diferente e necessita de ATP
Fatores que influenciam a atividade das enzimas de restrição
Composição do tampão
- Diferem na força iónica (concentração de sal);
- Diferem no catião principal (Na+ ou K+);
- Solução: Manter o pH da reação;
Temperatura de incubação
- A temperatura ótima da maioria das enzimas é de 37 ºC;
Exceções: Bactérias termofílicas – temperatura ótima de clivagem varia entre 50 a 60 ºC;
Influência da metilação no DNA
- Existe algumas endonucleases de restrição que não clivam DNA metilado;
Digestão com múltiplas enzimas
Existem enumeras bases de dados , onde é possível consultar informação sobre enzimas de restrição de restrição e os seus locais de reconhecimento. A REBASE® The Restriction Enzyme Database (http://rebase.neb.com) é uma ferramenta muito útil, especialmente desenvolvida para uso em biologia molecular e bioinformática.
References:
- Kulg; Cunnings (1997). Concepts of Genetics 5th ed., Internacional edition, Prentice Hall, USA.
- Watson, James D. (1992). Molecular Biology of the Gene 3rd ed., Benjamin/Lumming, Harvard University and Cold spring Haba laboratory.